Faltungsstrukturen des SARS-CoV2-Genoms zeigen Angriffspunkte für Medikamente und geben Hinweise auf zukünftige Coronaviren
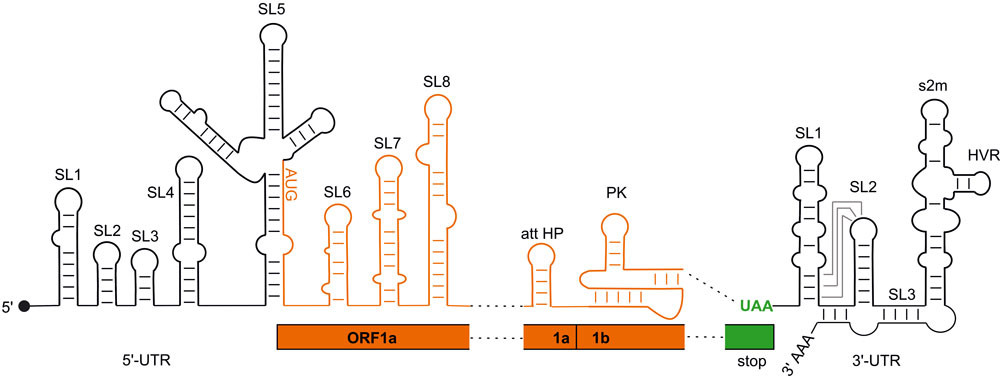
Regulatorische RNA-Elemente des SARS-CoV2-Genoms. Schwarz: Regionen, die nicht für Proteine kodieren (UTR). Orange: Regionen, die für Proteine kodieren (ORF).
Ein internationales Team unter Leitung von Wissenschaftler*innen der Rhein-Main-Universitäten (RMU), Chemiker*innen und Biochemiker*innen der Goethe-Universität Frankfurt und der TU Darmstadt, hat erstmals experimentell RNA-Faltungsstrukturen des SARS-CoV2-Genoms beobachtet. Mit den Faltungsstrukturen steuert das Virus den Infektionsverlauf. Da diese Strukturen bei verschiedenen Beta-Coronaviren sehr ähnlich sind, legen die Wissenschaftler*innen damit nicht nur die Grundlagen für die gezielte Entwicklung neuartiger Medikamente zur COVID-19-Behandlung, sondern auch für künftige Infektionsgeschehen mit neuen Coronaviren, die sich in der Zukunft entwickeln könnten.
Der Koordinator des Konsortiums, Prof. Harald Schwalbe vom Zentrum für Biomolekulare Magnetische Resonanz der Goethe-Universität Frankfurt, erläutert: „Mit unseren Ergebnissen haben wir eine breite Basis gelegt, um künftig genau zu verstehen, wie SARS-CoV2 das Infektionsgeschehen steuert. Wissenschaftlich war das ein gewaltiger, sehr arbeitsintensiver Kraftakt, den wir nur durch den außergewöhnlichen Einsatz der Teams hier in Frankfurt und Darmstadt gemeinsam mit unseren Partnern im COVID-19-NMR-Konsortium stemmen konnten. Doch es geht direkt weiter: Derzeit untersuchen wir zusammen mit unseren Kooperationspartnern, welche viralen Proteine und welche Proteine der menschlichen Wirtszellen mit den gefalteten regulatorischen Regionen der RNA interagieren, und ob sich daraus Ansatzpunkte für Therapien ergeben können.“
Ausführliche Informationen finden Sie hier.